Illumina Stranded mRNA Prep
Illumina Stranded mRNA Prep
Простое, универсальное, экономичное, быстрое решение для анализа кодирующего транскриптома за один день с использованием всего лишь 25 нг стандартной (не деградированной) РНК.
Illumina Stranded mRNA Prep предлагает оптимизированное решение RNA-Seq для точного и всестороннего анализа транскриптома. Набор подходит для различных типов образцов и поддерживает низкое количество стартового материала, вплоть до 25 нг РНК. Illumina Stranded mRNA Prep позволяет точно определять ориентацию оригинальной цепи, обеспечивает равномерное покрытие и достоверно обнаруживает такие признаки, как новые изоформы, фьюжены генов и аллель-специфическую экспрессию.
Исключительная производительность
Illumina Stranded mRNA Prep эффективно обеспечивает захват polyА и равномерность покрытия. Это минимизирует требуемую глубину секвенирования для точного обнаружения кодирующего транскриптома. Illumina Stranded mRNA Prep основан на технологии лигирования TruSeq, на которую ссылались в более чем 9 926 публикациях с 2011 года.
Универсальность для различных исследований
Illumina Stranded total RNA Prep может работать с разным количеством стартового материала РНК: от 25 нг до 1000 нг (не совместим с образцами FFPE).
Точное определение ориентации цепи
Эта информация позволяет обнаруживать антисмысловую транскрипцию, улучшает аннотацию транскрипта и повышает эффективность выравнивания.
Быстрый, удобный для автоматизации рабочий процесс
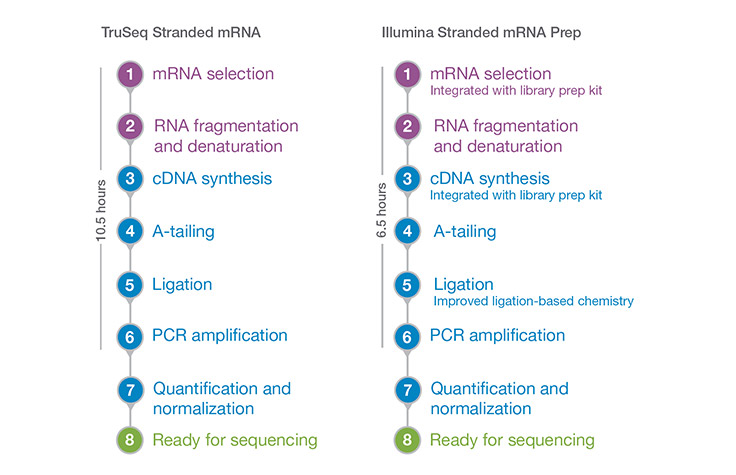
Подготовка библиотек для секвенирования занимает менее 7 часов. Методы автоматизации, разработанные партнёрами и признанные компанией Illumina, станут доступны в ближайшее время.
Универсальное секвенирование
Мультиплексирование до 384 образцов и секвенирование за один запуск. (Индексные наборы A и B уже доступны; наборы C и D скоро появятся).
Возможности RNA-Seq
RNA-Seq становится все более популярным, предоставляя подробный вид транскриптома в данный момент времени. Он предлагает многочисленные преимущества по сравнению с количественной ПЦР, в том числе:
- Экспериментальный дизайн без гипотез, не требующий предварительного знания транскриптома
- Больше возможностей для обнаружения известных и новых транскриптов
- Более высокая производительность для количественной оценки от сотен до тысяч участков в каждом анализе
- Более широкий динамический диапазон, обеспечивающий более точное измерение экспрессии генов
- Больше данных для анализа, предоставляющих полную информацию о последовательности и вариантах
Спецификации
|
Время анализа |
6,5 часов |
|---|---|
|
Время ручной работы |
< 3 часов |
| Тип нуклеиновой кислоты | РНК |
| Количество стартового материала |
25-1000 нг РНК стандартного качества |
| Особенности содержимого |
Захватывает кодирующий транскриптом с информацией о цепи |
|
Описание |
Простое, экономически эффективное решение для анализа кодирующего транскриптома с точной информацией о цепи |
| Мультиплексирование | До 384 уникальных двойных индексов (UDI) |
| Механизм действия |
Захват PolyA, добавление адаптеров и индексов на основе лигирования |
|
Для исследования каких видов подходит |
Мышь, Человек, Крыса, Млекопитающее, КРС |
| Виды. Примечание | Работает с высококачественной РНК любых видов с polyА хвостами |
| Метод |
Секвенирование mRNA |
|
Класс вариантов |
Фьюжены генов, новые транскрипты, однонуклеотидные полиморфизмы (SNP), варианты транскриптов |
| Совместимость | Набор совместим со всеми системами секвенирования Illumina, и тщательно проверен на системах NextSeq 500/550 и NovaSeq 6000 |
| Для работы с какими платформами подходит |
HiSeq 2500, HiSeq 3000, HiSeq 4000, NextSeq 2000, NextSeq 500, NextSeq 550, NovaSeq 6000 |
| Особые типы образцов | низкое количество материала образца, не подходит для работы с FFPE |
|
Стренд-специфичность набора |
Специфичен |
| Технология | Секвенирование |
| Возможность автоматизации | Автоматические станции пипетирования |
Рекомендации по проекту
|
Секвенатор |
Рекомендуемое количество образцов | Длина прочтения |
|---|---|---|
| NextSeq 550 |
Mid output: 5 High output: 16 (из расчета 25 миллионов прочтений на образец) |
2 х 75 п.о. |
| NextSeq 2000 |
P2: 16 P3: 40 (из расчета 25 миллионов прочтений на образец) |
2 х 75 п.о. |
| NovaSeq 6000 |
SP: 32 S1: 64 S2: 164 S4: 384 (из расчета 25 миллионов прочтений на образец) |
2 х 75 п.о. |
Сравнение с другими наборами
|
|
Illumina Stranded mRNA Prep |
Illumina Stranded Total RNA Prep with Ribo-Zero Plus |
Illumina RNA Prep with Enrichment |
TruSeq Stranded mRNA |
TruSeq RNA Library Prep Kit v2 |
|---|---|---|---|---|---|
|
Время анализа |
6,5 часов |
~7 часов |
< 9 часов | ~10,5 часов | 11,5 часов |
|
Возможность автоматизации |
Автоматические станции пипетирования |
Автоматические станции пипетирования |
Автоматические станции пипетирования |
Автоматические станции пипетирования |
Автоматические станции пипетирования |
|
Особенности содержимого |
Захватывает кодирующий транскриптом с информацией о нитях |
Захватывает кодирующую РНК плюс несколько форм некодирующей РНК |
Захватывает кодирующий транскриптом при использовании с Illumina Exome Panel |
Захватывает кодирующий транскриптом с информацией о цепи |
Захватывает кодирующий транскриптом (без информации о цепи) |
| Описание |
Простое, экономически эффективное решение для анализа кодирующего транскриптома с точной информацией о цепи |
Для секвенирования полного транскриптома нескольких типов образцов. Включает в себя модуль Ribo-Zero plus для удаления излишних транскриптов у нескольких видов |
Экономичное решение для обнаружения таргетных транскриптов из широкого спектра типов образцов, включая ткани FFPE и другие образцы низкого качества. |
Предоставляет исследователям четкое и полное представление о кодирующем транскриптоме с точной информацией о цепи. |
Простое, экономически эффективное исследовательское решение для анализа кодирующего транскриптома. |
|
Время ручной работы |
< 3 часов | < 3 часов | < 2 часов | ~4,5 часа |
~4,5 часа |
| Количество стартового материала | 25-1000 нг РНК стандартного качества | 1-1000 нг РНК для образцов стандартного качества. Рекомендуется минимум 10 нг РНК для оптимального результата и образцов FFPE | 10 нг РНК из свежих/замороженных образцов или 20 нг РНК из образцов FFPE |
0,1-1 мкг РНК или 10-100 нг ранее выделенной мРНК (из видов с polyA хвостами) |
0,1-1 мкг РНК или 10-400 нг ранее выделенной мРНК (из видов с polyA хвостами) |
| Механизм действия |
Захват PolyA, добавление адаптеров и индексов на основе лигирования |
Ферментативное удаление рРНК, добавление адаптеров и индексов на основе лигирования |
Bead-linked transposome |
Захват polyA хвостов посредством Oligo-dT на основе шариков |
Захват polyA хвостов посредством Oligo-dT на основе шариков |
| Мультиплексирование | До 384 уникальных двойных индексов (UDI) | До 384 уникальных двойных индексов (UDI) | До 384 уникальных двойных индексов (UDI) | 1-96 |
До 24 на дорожку |
|
Стренд-специфичность набора |
Специфичен |
Специфичен |
Не специфичен | Специфичен | Не специфичен |
|
Класс вариантов |
Фьюжены генов, новые транскрипты, однонуклеотидные полиморфизмы (SNP), варианты транскриптов |
Фьюжены генов, новые транскрипты, однонуклеотидные полиморфизмы (SNP), варианты транскриптов |
Фьюжены генов, новые транскрипты, однонуклеотидные полиморфизмы (SNP), варианты транскриптов | Фьюжены генов, новые транскрипты, однонуклеотидные полиморфизмы (SNP), варианты транскриптов | Фьюжены генов, новые транскрипты, однонуклеотидные полиморфизмы (SNP), варианты транскриптов |
| Технология | Секвенирование | Секвенирование | Секвенирование | Секвенирование | Секвенирование |
Вспомогательные данные и цифры
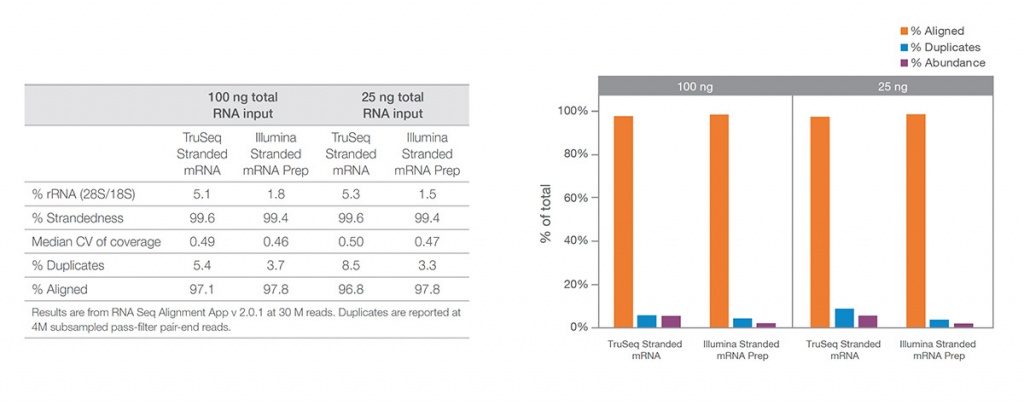
Сравнение показателей эффективности
Слева: показатели эффективности для Illumina Stranded mRNA Prep. Справа: Illumina Stranded mRNA Prep сравнивали с TruSeq Stranded mRNA. Illumina Stranded mRNA Prep показал превосходящую эффективность, особенно при вводе 25 нг универсальной человеческой эталонной РНК. Библиотеки имели до 30 млн. прочтений на образец и были проанализированы с помощью приложения BaseSpace RNA-Seq Alignment v2.0.1. Дупликаты выявляются на подвыборке из 4 млн парных ридов, прошедших фильтр.
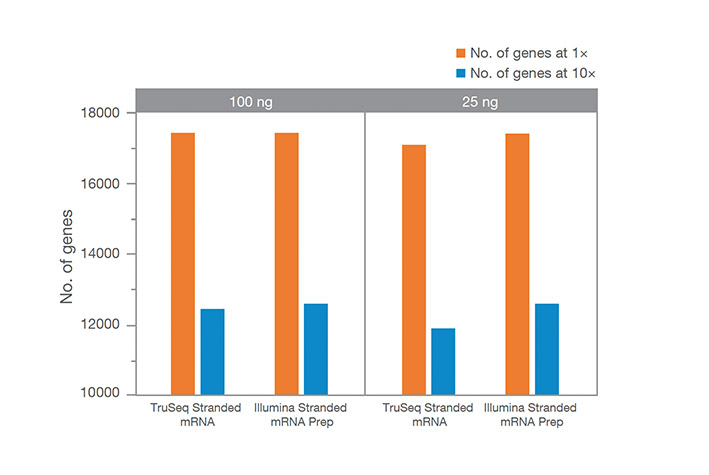
Точное обнаружение гена при малом количестве стартового материала
Illumina Stranded mRNA Prep обеспечивает точное обнаружение генов при малом количестве РНК по сравнению с TruSeq Stranded mRNA. Количество обнаруженных генов регистрируется при 30 млн. парно-концевых чтений на образец при прохождении фильтров PF. Детектирование большего числа генов при 1х покрытии в случае применения Illumina Stranded mRNA Prep является индикатором большей чувствительности данного набора.
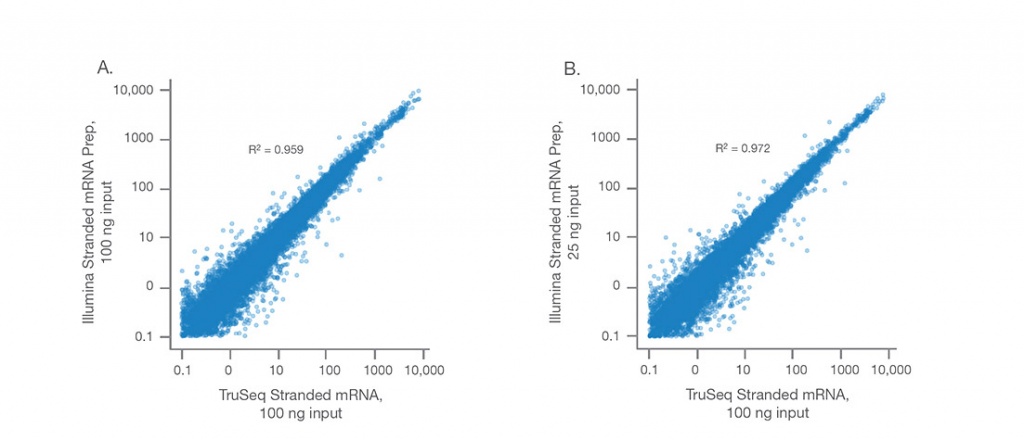
Высокое соответствие с Legacy Kit
Illumina Stranded mRNA Prep обеспечивает высоко согласованные данные с TruSeq Stranded mRNA при (A) одинаковом количестве стартового материала - 100 нг универсальной человеческой эталонной РНК и (B) с меньшим количеством стартового материала - 25 нг в сравнении с 100 нг универсальной человеческой эталонной РНК.
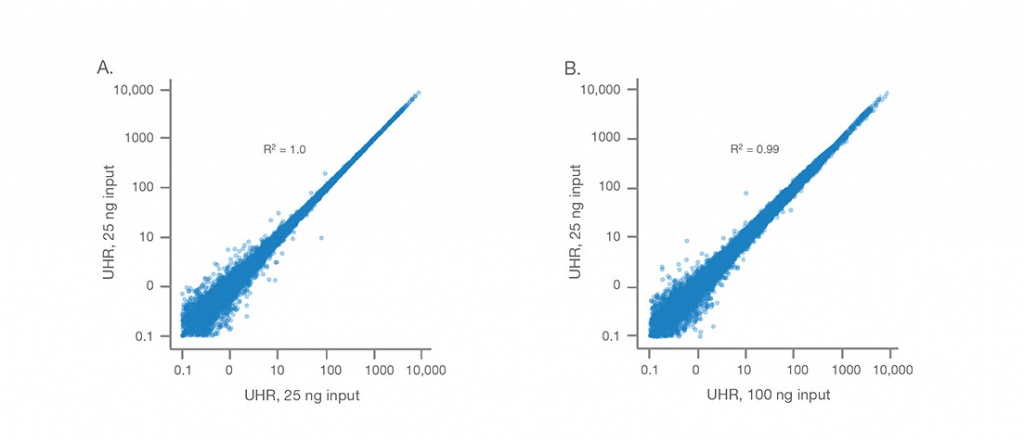
Высокая согласованность данных
Illumina Stranded mRNA Prep обеспечивает высокую согласованность данных между (A) техническими репликами 25 нг универсальной человеческой эталонной РНК и (B) между образцами универсальной человеческой эталонной РНК со стартовым количеством в 25 нг и 100 нг . Библиотеки секвенировали в режиме 2 × 74 п.о., при 30 млн. прочтений на образец. Анализ данных проводился с использованием приложения BaseSpace RNA-Seq Alignment v2.0.1.
Рабочий процесс Illumina Stranded mRNA Prep Workflow
Illumina Stranded mRNA Prep обеспечивает быстрый рабочий процесс с уменьшенным временем работы руками. Время может варьироваться в зависимости от используемого оборудования, количества обрабатываемых образцов, процедур автоматизации или опыта пользователя.